-
Hierarchy Clustering(계층적 군집화) 및 Dendrogram Visualization(덴드로그램 시각화) - AgglomerativeClustering, linkage, dendrogramData Science/ML&DL 모델 2022. 10. 5. 20:40반응형
해당 글에서는 hierarchy clustering(계층적 클러스터링,군집화)을 파이썬으로 구현해본다.
사용되는 라이브러리는 아래와 같다.
모델 : scipy.cluster.AgglomerativeClustering
거리계산 : scipy.cluster.hierarchy.linkage
시각화 : scipy.cluster.hierarchy.dendrogram
평가 : sklearn.metrics.silhouette_score , silhouette_samples, yellowbrick.cluster.KElbowVisualizer
Clustering에 대한 이론적인 부분은 "클러스터링 기법(개념,타당성,평가)" 글을 참고.
1. 데이터 준비
sklearn에서 제공해주는 iris 데이터를 사용한다.
clustering이기에 종속변수를 제외하고 독립변수만으로 데이터프레임을 구성한다.
import pandas as pd from sklearn import datasets iris = datasets.load_iris() X = iris.data y = iris.target df = pd.DataFrame(X, columns = iris.feature_names) print(df)
clustering에서는 각 독립변수(컬럼)별로 값의 스케일이 다르면 가지게되는 가중치가 달라진다.
( 독립변수 x1이 1000~10000의 값을 가지고 독립변수 x2가 1~3의 스케일을 가질경우 x1의 변화에 따라서 모델이 좌우되고 x2의 변화는 무시된다.)
따라서 스케일링을 통해서 모든 독립변수간의 스케일을 동일하게 맞추어준다.
from sklearn.preprocessing import StandardScaler scaler = StandardScaler() df.loc[:,:] = scaler.fit_transform(df) print(df)
2. AgglomerativeClustering 생성 및 학습
sklearn의 모든 모델들과 같이 .fit명령어로 학습한다.
clustering은 비지도 학습이므로 독립변수만을 입력으로 받는다.
from sklearn.cluster import KMeans kmeans = KMeans(n_clusters=3, random_state=7) kmeans.fit(df)파라미터 기능 초기값 파라미터 n_clusters cluster 개수 지정 입력필수 정수값 random_state randomseed 값 None 정수값 linkage distance 측정 방법 지정 ward ‘ward’, ‘complete’, ‘average’, ‘single’ 분류 결과는 .labels_로 확인가능하다.
agg.labels_
Hierarchy Clustering은 군집 중심을 정한것이 아닌, 제일 가까운 거리끼리 군집하는 방법으로 새로운 데이터에대한 transform 명령어가 없다.
3. Dendrogram Visualization(덴드로그램 시각화)
linkage 라이브러리 및 dendrogram 라이브러리를 사용해서 Hierarchy Clustering을 시각화 할 수 있다.
from scipy.cluster.hierarchy import dendrogram, linkage Z = linkage(df, method="ward") fig = plt.figure(figsize=(10, 6)) dendrogram(Z, labels=y) plt.title('Hierarchical Clustering Dendrogram (ward linkage)') plt.xlabel('Company') plt.show()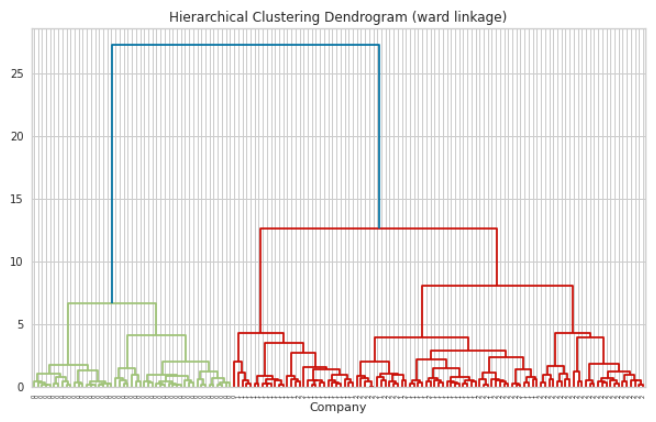
4. 타당성 평가(성능 평가)
Clustering은 정답이 없는 비지도학습이기에 모델의 타당성(또는 성능)을 평가하기위해서 거리값을 사용한다.
또한, cluster 개수별로 타당성을 비교해서 최적의 cluster 개수를 구하는데 사용한다.
4.1 Elbow method(엘보우 기법)
군집 내 오차제곱합(SSE, sum of squared error)의 합을 군집화의 비용함수로 정의.
군집 수가 늘어날수록 비용함수는 감소함.
이때, 비용함수 감소 기울기가 급격히 줄어드는 부분(더이상 군집을 늘려도 크게 비용이 감소하지 않는 부분)을 최적의 군집수로 판단한다.
KElbowVisualizer 라이브러리를 활용하여 손쉽게 계산 및 시각화를 할 수 있다.
from yellowbrick.cluster import KElbowVisualizer k=0 agg = AgglomerativeClustering(n_clusters=k) visualizer = KElbowVisualizer(agg, k=(1,6), timings=False) visualizer.fit(df) visualizer.show()
위 그림에서 군집수 2개가 가장 최상의 군집 개수라고 추천하고 있다.
4.2 Silhouette Index(실루엣 인덱스)
clustering 기법에서 실질적으로 가장 많이 쓰이는 타당성(평가) 지표이다.
개별객체마다 silhouette index 값을 구할수 있으며, 이에대한 평균값을 사용한다.
모든값이 0.5 이상을 가지면 유의하다고 판단한다.
a(i) : 객체 i와 객체 i가 속한 cluster 내 다른 객체들간의 거리 평균
b(i) : 객체 i와 다른 군집에 속한 객체들간의 거리평균 중, 가장 최소값을 가지는 군집과의 거리 평균
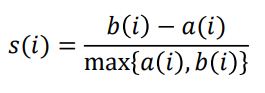
from sklearn.metrics import silhouette_score, silhouette_samples silhouette_avg = silhouette_score(df, kmeans.labels_) print(silhouette_avg) #=>0.33058726295230545cluster 개수별로 silhouette index를 계산하여서 가장높은 값을 가지는 cluster 수를 최적의 값으로 본다.
for k in range(2, 6): agg = AgglomerativeClustering(n_clusters=k).fit(df) silhouette_avg = silhouette_score(df, agg.labels_) print(f'cluster : {k} // silhouette index {silhouette_avg}')
위의 결과에서는 cluster 2개에서 가장 높은 index 값을 가졌으므로 2가 가장 유의한 cluster 개수라고 할 수 있다.
이에대한 시각화를 추가하면 아래와 같다.
import matplotlib.pyplot as plt import numpy as np for k in range(2, 6): fig, ax = plt.subplots(1, 1,figsize=(4,3)) agg = AgglomerativeClustering(n_clusters=k).fit(df) silhouette_vals = silhouette_samples(df, agg.labels_) y_ticks = [] y_lower = y_upper = 0 for c_num in np.unique(agg.labels_): cluster_silhouette_vals = silhouette_vals[agg.labels_==c_num] y_upper += len(cluster_silhouette_vals) cluster_silhouette_vals.sort() # numpy array는 sort 메소드를 이용하면 오름차순으로 정렬된 형태로 바뀜 ax.barh(range(y_lower, y_upper), cluster_silhouette_vals, height=1) y_lower += len(cluster_silhouette_vals) 반응형
반응형'Data Science > ML&DL 모델' 카테고리의 다른 글
AutoEncoder 개념 및 종류 (0) 2022.10.31 Transformer Model 개념 및 모델 구조 (0) 2022.10.28 서포트 벡터 머신(SVM,Support Vector Machine) 파이썬 구현하기(cvxpy, sklearn (0) 2022.10.14 DBSCAN(Density-based spatial clustering of applications with noise, 밀도기반 군집화) 파이썬구현하기 (0) 2022.10.05 K-means Clustering 파이썬 구현하기 (0) 2022.10.05